Werbung
Dr. Klaus Schulten von der University of Illinois stellt seine Arbeit im Zusammenhang mit der Entwicklung von CUDA vor. Seine alltägliche Arbeit beschäftigt sich mit Proteinen, im speziellen hat er sich mit der Resistenz von Tamiflu gegenüber der Schweinegrippe beschäftigt. Die Wissenschaftler konnten sich nicht erklären wie der Schweinegrippe-Erreger eine Resistenz gegenüber dem Protein in Tamiflu entwickeln konnte. Der klassische Ansatz sich das Protein durch ein Mikroskop anzuschauen, half den Wissenschaftlern nicht weiter und so entwickelten sie ein „Mikroskop“ aus chemischen, physikalischen und mathematischen Formeln, welche in einer Software zusammengeführt wurden.
{jphoto image=2763}
Diese Software sollte auf Supercomputern die nötigen Berechnungen ausführen. Bereits in der gestrigen Keynote stellte Jen-Hsun Huan Multi-GPU AMBER 11 vor, eine Software zur Simulation von Molekülen und deren Verhalten wenn zwei oder mehr miteinander reagieren. Hier erreichten acht Fermi-GPUs die Berechnung von 52 ns pro Tag. Ein Supercomputer mit 192 Quad-Core-CPUs erreichte 46 ns pro Tag. Eben diesen Umstand machte sich auch das Team von Dr. Klaus Schulten zu nutze und setzte bei der Berechnung des Tamiflu-Proteins welches auf den Schweinegrippe-Erreger reagieren sollte GPUs zur Verarbeitung der Daten ein. Dabei sollte auch erwähnt werden, dass hier nicht nur einige wenige Moleküle miteinander reagieren, sondern mehrere 100 Millionen Atom-Langzeit-Simulationen durchgeführt werden müssen.
Durch Klick auf das Bild gelangt man zu einer vergrößerten Ansicht
Was auf einem klassischen Supercomputer bereits etwa 90 Minuten dauert, konnte mit Hilfe von GPUs auf drei Minuten reduziert werden. Neben der Einsparung von Rechenzeit spielt auch der Stromverbrauch bzw. die Effektive Nutzung eine entscheidende Rolle. Eine Berechnung auf Basis von CPUs verbrauchte 448 Watt-Stunden, auf Basis von GPUs sind es 43 Watt-Stunden.
Durch Klick auf das Bild gelangt man zu einer vergrößerten Ansicht
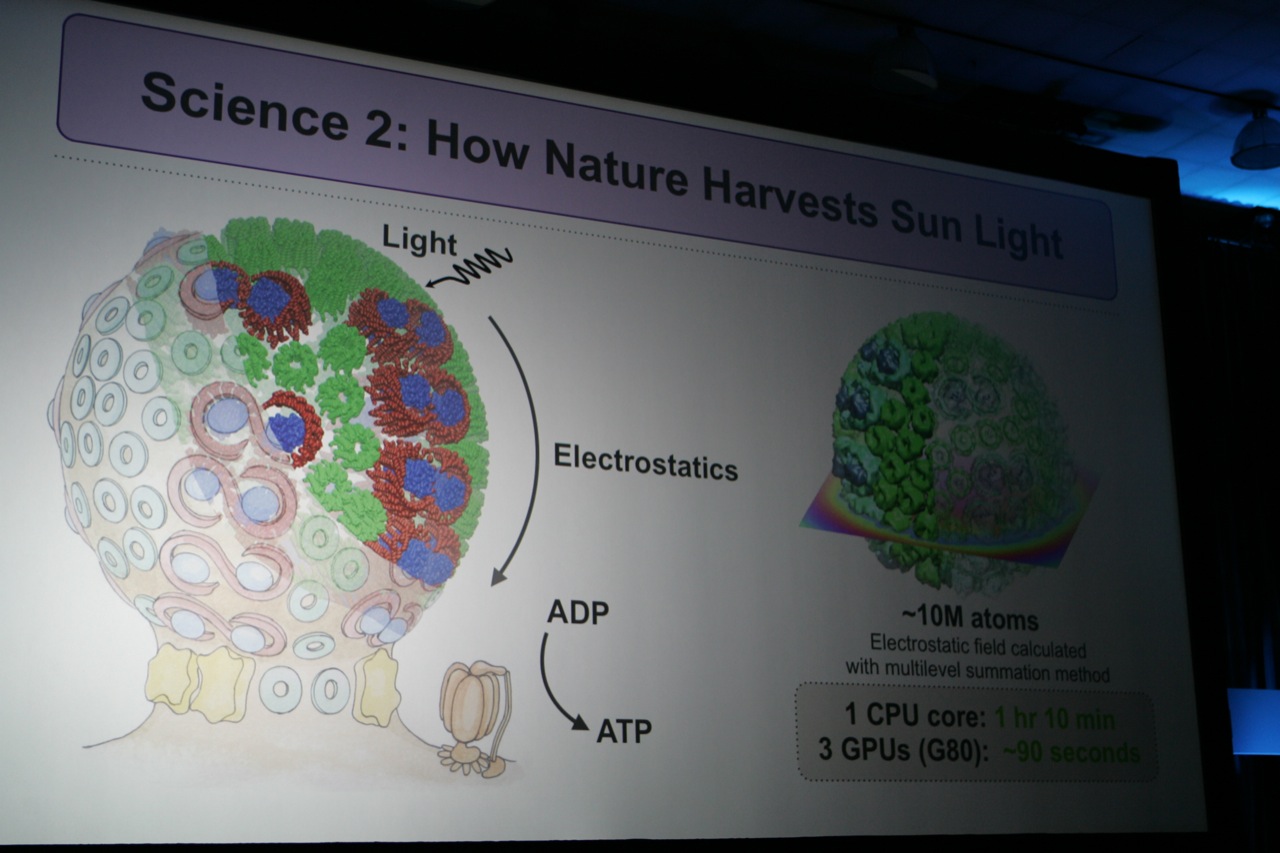
Neben der Untersuchung des Proteins von Tamiflu beschäftigt sich Dr. Klaus Schulten auch mit der Fotosynthese. 95% der Energie in der Biosphäre wird durch die Fotosynthese entwickelt. Noch immer aber verstehen die Wissenschaftler nicht, wie genau dieser Prozess abläuft. Das Verhalten bzw. das elektromagnetische Feld von 10 Millionen Atomen innerhalb einer Zelle muss dazu berechnet werden. Auf einer CPU dauerte dies 70 Minuten. Auf drei G80-GPUs konnte dies auf etwa 90 Sekunden reduziert werden.
Durch Klick auf das Bild gelangt man zu einer vergrößerten Ansicht
Ein drittens Anwendungsgebiet ist Entstehung eines Proteins aus RNA-Informationen. Für die Beantwortung dieser Frage erhielt ein Team von Wissenschaftlern 2009 den Nobel-Preis. Deren Forschung beschäftigte sich mit einem statischen Zustand. Viel wichtiger aber ist die Betrachtung des dynamischen Prozesses. Diese molekular-dynamische Simulation wurde auf dem NCSA Lincoln Cluser ausgeführt, welches über acht Intel Cores und zwei NVIDIA Tesla GPUs pro Rechenknoten verfügt. Der Einsatz von immer mehr GPUs pro Cluster hat die Rechenzeit dramatisch reduzieren können.
Durch Klick auf das Bild gelangt man zu einer vergrößerten Ansicht
Die vierte Anwendung von GPGPU beschäftigt sich ebenfalls mit der Interaktion von Atomen. Im Falle von DNA-Ketten muss das Verhalten von 4,7 Millionen Atomen berechnet werden. Auf einem Intel Xeon X5550 Quad-Core benötigte diese Berechnung 15 Stunden. Auf vier NVIDIA C2050 GPUs erfolgte die Verarbeitung in zehn Minuten. Dabei konnte man hier besonders von der Fermi-Architektur profitieren, die rund 3x schneller ist als Tesla.
Weitere Links: