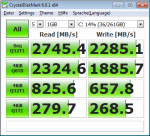
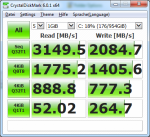
Das passt ja wie bestellt

. Genau vor diesem Problem stehe ich momentan auch (MS-core facility) und stehe ohne IT Support da.Wir machen alles, Elementaranalytik, Bioanalytik und Kleinstmoleküle. Nebenher auch noch HDX. Frage am Rande, arbeitet ihr nach DFG Leitlinien bezüglich Datenspeicherung? Und wenn dir das hier im Thread zu viel wird, gerne per PM oder Mail (offiziell von Institut zu Institut).
Bin nicht in der Privatwirtschaft (Pharma, Entwicklung mit Fokus auf Proteine und pDNA), also betrifft mich DFG nicht, aber wie Daten sicher gespeichert und auch in 25 Jahren noch auffindbar sind ist natürlich auch für uns ein großes Thema. (sind ja verpflichtet Rohdaten mindestens 25 Jahre zu speichern)
Details können wir bissl über PM austauschen, hier im Forum nicht im Detail, sonst bräuchte ich gleich mit allen ein CDA

Wir haben hier 6 Massenspektrometer, ein weiteres ist in der Beantragung. Ergo, wir setzen hier momentan 5 verschiedene Softwareprodukte für die Auswertung ein.
Datensätze liegen zwischen 1Gb-300Gb pro Experiment.
Bei unserem Bioinfomatikworkflow (Proteome Discoverer/PEAKS/MaxQuant wem es was sagt), dauert die Prozessierung einer Probe normalerweise 2h (Workstation ist ein Dual 6C/12T Xeon X5xxx, 64Gb RAM, RAID 0 für Rohdaten, SATA-SSD für Sequenzen), eine komplizierte Probe (Phospho-Enrichment) dauert 9h in Etwa.
Dann teilen wir uns zumindest ein Programm, nämlich den Thermo Proteome Discoverer, wir nutzen sonst noch Thermo Biopharma Finder.
Zusätzlich, als dritte SW beachte ich noch die SW auf die wir in Zukunft switchen wollen (Protein metrics), da haben wir die Evaluierung abgeschlossen und sind nun in Lizenzverhandlungen. (wer diese Preise einmal gesehen hat, ärgert sich nie wieder über Adobe-Preise

)
Generell ist die Thematik ja etwas komplexer, weil man soviel berücksichtigen muss.
Alleine die verwendete MS-Plattform liefert schon völlig unterschiedliche Datenmengen (bei uns Orbitrap und eine Xevo TQ-XS), dann kommt noch dazu dass natürlich die Settings der "Datenaufnahme" die Dateigröße und die Komplexität der nachträglich ausgeführten Analyse extrem starken Einfluss haben. D.h., wenn jemand in einem Paper etwas beschreibt kann man das nicht direkt umsetzen.
Ich sehe zB bei uns überhaupt keine Limitierung durch die HDDs, muss ich mir trotzdem überlegen ob ich nicht doch ein zweite SSD dazu konfiguriere. Bei Protein Metrics hat damals bei der Evaluierung darauf natürlich niemand geschaut und die Testversion ist bereits abgelaufen, d.h. da kann ich nichts mehr machen.
Ist das Windows oder Unix/Linux based? C:\ und MSSQL klingt klar nach Windows?
Bei mir auch nur Windows, mit der neuen Workstation Umstieg auf Windows 10 Pro Workstation
Weil wenn Windows könnte man doch für ne halbwegs brauchbare Aussage einfach mal A) ne Dual CPU Maschine nehmen (habt ihr ja offenbar beide so ein Teil) und dann B) die "Paralellisierung runterschrauben" - so dass die genutzte Threadanzahl maximal dem halben der gesamt verfügbaren Threads entspricht. Der Windows Taskmanager bspw. gibt dann auskunft, ....
Check ich, vielen Dank.
AVX lass ich mal links liegen, bei der Thermo-SW bin ich mir, sowie ich Thermo kenne, sicher dass die sich nie um optimierten Code gekümmert haben.
Bei Protein metrics habe ich nochmal angefragt, schauen wir mal was sie sagen.
Thema IO/Storage - wenn das storage lastig ist, bringt viel RAM meist ne Menge Punkte - Stichwort Disk-Cache. Ein modernes OS kippt dir so viel wie möglich Daten in den RAM und nutzt den Spaß als lokalen Cache. Das macht übel viel Speed aus. SSDs (ggf. gleich ne PCIe oder M.2 NVMe Version) würden das dann nochmals weiter beschleunigen.
Wie weiter oben geschrieben, ich sehe die IO-Lastigkeit bei unserem Workflow nicht so wie Storm, aber werd ich nochmal checken. Kommt halt eine SSD rein oder ähnliches noch zusätzlich rein für aktuelle Daten in Bearbeitung und das RAID 5 für Archivierung bis die Daten monatlich auf den Server gesichert werden.
Wir arbeiten auch nicht mit MASCOT oder ähnlichem, ich vermute mal stark dass die Datenbank bei Storm hier wirklich IO-lastig ist, wir sind nur sehr selten bei Identifizierungen mit Datenbanksuchen unterwegs.
Zum Konfigurator bei Lenovo - frag mal dort ob da noch was existiert. Wie gesagt, ich nutze hier Fujitsu - da kannst du Online was zusammen klickern -> das taugt meist aber nix. Es gibt aber ein Tool, da hast du völlig freie Hand. Das ist auch "public" - nur findest du es eben nicht groß und fett auf der Webseite verlinkt - man muss wissen das es das gibt und gezielt danach suchen -> dann bekommt man das auch. Keine Ahnung ob das bei Lenovo sowas auch gibt. Denke aber schon... Bei HP(E) gibts das, bei Fujitsu, bei Cisco (wenn dort auch als Web-Version) usw. Schwer vorstellbar, dass es da nur diesen rudimentären Webmist gibt...
Mach mich mal auf die Suche, derzeit ist Lenovo so lahm dass sie mir nach 3 Arbeitstagen noch nicht einmal ein Angebot für die 2 Konfigs geschickt haben, möchte nicht wissen wie lange dass dann bei denen dauert wenn man wirklich noch spezieller konfiguriert.
Wie viel schneller muss es denn werden?
Und wie viel teurer als die Hardware ist die Softwarelizenz?
Einfach gesagt, die beiden aktuellen SW halten sich von den Preisen her in Grenzen, jene welche in Evaluierung ist kostet eine Lawine, bei weitem mehr pro Jahr als die Investition in die Workstation
Ich bin in diesem Fall ja sogar in dem Vorteil dass es das Budget meiner Laborgruppe ist, das heißt ich brauch (bis zu einem bestimmten Limit niemand wegen dem Geld fragen), mein Scientist für die Masse ist aber kein IT-Nerd, sondern MS-Nerd

Darum wollte ich die Konfig für Ihn erledigen, naja, lern ich was dazu.

Aber vielen Dank auf jeden Fall schon mal vorab für all die guten Hinweise bisher, ich werd mir auch mal anschauen wo ich mit einer Single-CPU Workstation mit einer 20-Core CPU preislich lande, vielleicht doch eine interessante Option.


 ) zurückgreifen muss.
) zurückgreifen muss.